

DeCodi-Fi™
Polimerasa de Alta Fidelidad
PARA AMPLIFICACIÓN DE FRAGMENTOS DE ADN LARGOS


DeCodi-Fi™
Polimerasa de Alta Fidelidad
PARA AMPLIFICACIÓN DE FRAGMENTOS DE ADN LARGOS

Llega más lejos.
Explora el genoma completo con DeCodi-Fi™.
DeCodi-Fi™ es una polimerasa de ADN de alta fidelidad diseñada para amplificar fragmentos largos de ADN, de hasta 44 kb, con una fidelidad excepcional, alto rendimiento, ciclos rápidos y un desempeño robusto incluso a partir de cantidades ultra bajas de ADN.
Optimizada para aplicaciones de vanguardia —como la secuenciación de lectura larga, el ensamblaje de genomas completos y la detección de variantes estructurales— proporciona la longitud y la precisión necesarias incluso cuando la cantidad de ADN es limitada, cerrando la brecha entre bajo input y éxito en amplificaciones de largo alcance.

Amplifica más.
Explora el genoma completo con DeCodi-Fi™.
DeCodi-Fi™ es una polimerasa de ADN de alta fidelidad capaz de amplificar fragmentos largos, hasta 44 kb, con alta precisión y rendimiento incluso desde cantidades mínimas de ADN.
Diseñada para secuenciación de lectura larga, ensamblaje de genomas y detección de variantes estructurales, ofrece la longitud y exactitud necesarias aun con bajo input.
-
Fragmentos más largos: Amplifica hasta 44 kb, generando amplicones de alto rendimiento para plataformas de secuenciación de lectura larga.
-
Compatibilidad con bajo input: Amplifica ADN complejo desde 0,1 ng y fragmentos de complejidad media desde 5 pg.
-
Alta fidelidad: Más de un 60% de reducción en la tasa de errores, logrando una amplificación de alta precisión para aplicaciones donde la exactitud es crítica.
-
Alto rendimiento, bajo sesgo: Amplificación uniforme y alta recuperación de ADN, incluso a partir de fragmentos desafiantes o ricos en GC (25%–85%).
-
Ciclos rápidos: Logra una amplificación rápida, inferior a 15 s/kb.
-
Compatible con uracilo para aplicaciones epigenéticas: Amplifica eficientemente fragmentos con uracilo, compatible con análisis de metilación y ADN dañado.
-
Amplificación de fragmentos de ADN para clonación
-
Amplificación de fragmentos largos o ricos en GC
-
Amplificación de bibliotecas para secuenciación
-
PCR de alta fidelidad y especificidad
-
PCR de alto rendimiento (High-throughput PCR)
Technical Data Sheets
![]() DeCodi-Fi All-In-One Mix TDS / Protocol
DeCodi-Fi All-In-One Mix TDS / Protocol
![]() DeCodi-Fi PCR Kit TDS / Protocol
DeCodi-Fi PCR Kit TDS / Protocol
Brochure
![]() DeCodi-Fi High-Fidelity Polymerase Brochure
DeCodi-Fi High-Fidelity Polymerase Brochure
Posters
![]() Unveiling Microbial Diversity Cost-Effective 16S rRNA Amplicon Sequencing using DeCodi-Fi High Fidelity Polymerase
Unveiling Microbial Diversity Cost-Effective 16S rRNA Amplicon Sequencing using DeCodi-Fi High Fidelity Polymerase
![]() DeCodi-Fi: A Cost-Effective Polymerase for High-Quality Genome Assembly of Marine Sediment Bacteria
DeCodi-Fi: A Cost-Effective Polymerase for High-Quality Genome Assembly of Marine Sediment Bacteria
![]() Solving amplification of high-GC expanded repeats to enable rare disease detection using long-read sequencing
Solving amplification of high-GC expanded repeats to enable rare disease detection using long-read sequencing
![]() A long-read 16S rRNA sequencing workflow enhances bacterial diversity recovery from rhizospheric soils of a salt flat in the Atacama desert
A long-read 16S rRNA sequencing workflow enhances bacterial diversity recovery from rhizospheric soils of a salt flat in the Atacama desert
Application Notes
![]() Resistance of High-Fidelity DNA polymerases to magnetic beads' presence
Resistance of High-Fidelity DNA polymerases to magnetic beads' presence
![]() Direct PCR from Crude Plant Extracts Using DeCodi-Fi High-Fidelity Polymerase
Direct PCR from Crude Plant Extracts Using DeCodi-Fi High-Fidelity Polymerase
![]() DeCodi-Fi in Amplicon Sequencing: A Sensitive High-Fidelity Polymerase for Xanthomonas citri Pathovar Differentiation
DeCodi-Fi in Amplicon Sequencing: A Sensitive High-Fidelity Polymerase for Xanthomonas citri Pathovar Differentiation
![]() Oxford Nanopore 16S Sequencing of Soil Samples from the Atacama Desert
Oxford Nanopore 16S Sequencing of Soil Samples from the Atacama Desert
![]() Comparative analysis of size bias in PCR Multiplex amplification using DeCodi-Fi™ vs commercial HiFi polymerases
Comparative analysis of size bias in PCR Multiplex amplification using DeCodi-Fi™ vs commercial HiFi polymerases
Quick Start Guides
![]() DeCodi-Fi All-In-One Mix Quick Start Guide
DeCodi-Fi All-In-One Mix Quick Start Guide
![]() DeCodi-Fi PCR Kit Quick Start Guide
DeCodi-Fi PCR Kit Quick Start Guide
-
Fragmentos más largos: Amplifica hasta 44 kb, generando amplicones de alto rendimiento listos para plataformas de secuenciación de lectura larga.
-
Compatibilidad con bajo input: Amplifica ADN complejo desde solo 0,1 ng y plantillas de complejidad media desde 5 pg.
-
Alta fidelidad: 64% mayor fidelidad que KAPA HiFi, reduciendo las tasas de error en aplicaciones de alta precisión.
-
Alto rendimiento, bajo sesgo: Amplificación uniforme y alta recuperación de ADN, incluso a partir de plantillas exigentes o ricas en GC (25%–85%).
-
Ciclos rápidos: Logra una amplificación veloz con velocidades de extensión inferiores a 15 s/kb.
-
Compatible con uracilo para flujos epigenéticos: Tolera uracilo, permitiendo estudios de metilación y protocolos tolerantes al daño.
-
Amplificación de fragmentos de ADN para clonación
-
Amplificación de fragmentos largos o ricos en GC
-
Amplificación de bibliotecas para secuenciación
-
PCR de alta fidelidad y especificidad
-
PCR de alto rendimiento (High-throughput PCR)
Technical Data Sheets
![]() DeCodi-Fi All-In-One Mix TDS / Protocol
DeCodi-Fi All-In-One Mix TDS / Protocol
![]() DeCodi-Fi PCR Kit TDS / Protocol
DeCodi-Fi PCR Kit TDS / Protocol
Brochure
![]() DeCodi-Fi High-Fidelity Polymerase Brochure
DeCodi-Fi High-Fidelity Polymerase Brochure
Posters
![]() Unveiling Microbial Diversity Cost-Effective 16S rRNA Amplicon Sequencing using DeCodi-Fi High Fidelity Polymerase
Unveiling Microbial Diversity Cost-Effective 16S rRNA Amplicon Sequencing using DeCodi-Fi High Fidelity Polymerase
![]() DeCodi-Fi: A Cost-Effective Polymerase for High-Quality Genome Assembly of Marine Sediment Bacteria
DeCodi-Fi: A Cost-Effective Polymerase for High-Quality Genome Assembly of Marine Sediment Bacteria
![]() Solving amplification of high-GC expanded repeats to enable rare disease detection using long-read sequencing
Solving amplification of high-GC expanded repeats to enable rare disease detection using long-read sequencing
![]() A long-read 16S rRNA sequencing workflow enhances bacterial diversity recovery from rhizospheric soils of a salt flat in the Atacama desert
A long-read 16S rRNA sequencing workflow enhances bacterial diversity recovery from rhizospheric soils of a salt flat in the Atacama desert
Application Notes
![]() Resistance of High-Fidelity DNA polymerases to magnetic beads' presence
Resistance of High-Fidelity DNA polymerases to magnetic beads' presence
![]() Direct PCR from Crude Plant Extracts Using DeCodi-Fi High-Fidelity Polymerase
Direct PCR from Crude Plant Extracts Using DeCodi-Fi High-Fidelity Polymerase
![]() DeCodi-Fi in Amplicon Sequencing: A Sensitive High-Fidelity Polymerase for Xanthomonas citri Pathovar Differentiation
DeCodi-Fi in Amplicon Sequencing: A Sensitive High-Fidelity Polymerase for Xanthomonas citri Pathovar Differentiation
![]() Oxford Nanopore 16S Sequencing of Soil Samples from the Atacama Desert
Oxford Nanopore 16S Sequencing of Soil Samples from the Atacama Desert
![]() Comparative analysis of size bias in PCR Multiplex amplification using DeCodi-Fi™ vs commercial HiFi polymerases
Comparative analysis of size bias in PCR Multiplex amplification using DeCodi-Fi™ vs commercial HiFi polymerases
Quick Start Guides
LA POLIMERASA DE ALTA FIDELIDAD MÁS ACCESIBLE DEL MERCADO
DeCodi-Fi™ combina alto rendimiento con una excelente relación costo-beneficio, ofreciendo resultados de calidad superior a un precio competitivo.
LA POLIMERASA DE ALTA FIDELIDAD MÁS ACCESIBLE DEL MERCADO
DeCodi-Fi™ combina alto rendimiento con una excelente relación costo-beneficio, ofreciendo resultados de calidad superior a un precio competitivo.
DATOS DE RENDIMIENTO
AMPLIFICACIÓN LARGA Y ALTO RENDIMIENTO +
Maximiza el rendimiento del fragmento objetivo con una amplificación altamente eficiente, incluso en fragmentos de hasta 44 kb de longitud.
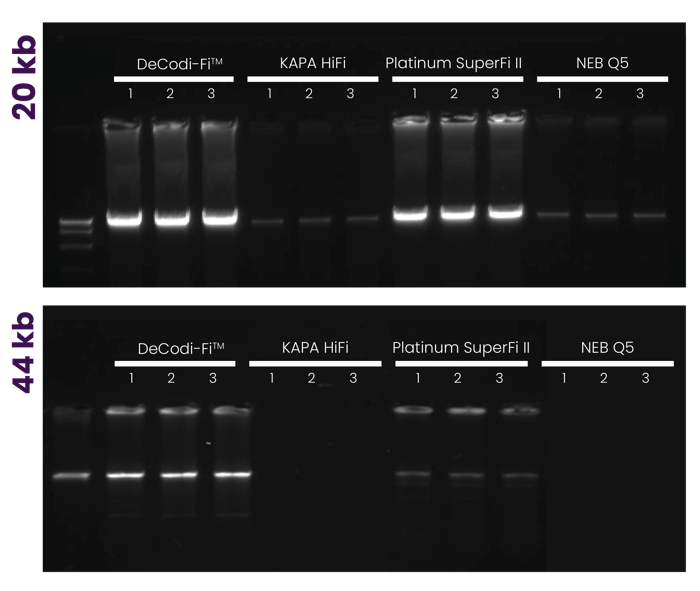
Se realizó la amplificación de fragmentos de ADN lambda de 20 kb y 44 kb con DeCodi-Fi™ High-Fidelity Polymerase, KAPA HiFi, Platinum SuperFi II y Q5. Cada fragmento se amplificó a partir de una baja cantidad de ADN (1 ng). Las reacciones se llevaron a cabo bajo condiciones optimizadas para cada enzima, utilizando 24 ciclos. El experimento se realizó por triplicado.
BAJO INPUT +
Lleva la secuenciación de lectura larga al siguiente nivel incluso con cantidades mínimas de muestra. DeCodi-Fi™ amplifica fragmentos largos a partir de solo 5 pg, mientras que otros métodos no logran resultados ni siquiera con 50 pg.

La amplificación del gen humano de beta-globina de 17,6 kb se realizó utilizando DeCodi-Fi™ High-Fidelity Polymerase y KOD Xtreme™ Hot Start DNA Polymerase. Cada blanco se amplificó a partir de diferentes cantidades de ADN en reacciones de 25 μL: 1000 pg, 125 pg y 50 pg. También se incluyó un control sin plantilla (0 pg). Todas las reacciones se llevaron a cabo siguiendo las condiciones optimizadas para cada enzima y utilizando 30 ciclos. El experimento se realizó por triplicado.
ALTA FIDELIDAD +
DeCodi-Fi™ High-Fidelity Polymerase muestra una reducción del 64% en la tasa de errores en comparación con KAPA HiFi.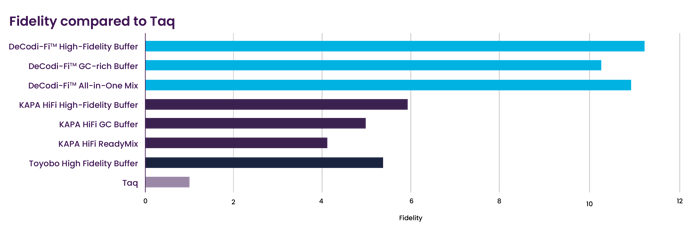
Comparación de DeCodi-Fi™, KAPA HiFi y Toyobo HiFi, medida en términos de mejora relativa sobre Taq Polymerase, basada en bibliotecas de Illumina del genoma completo de E. coli.
ALTO RENDIMIENTO +
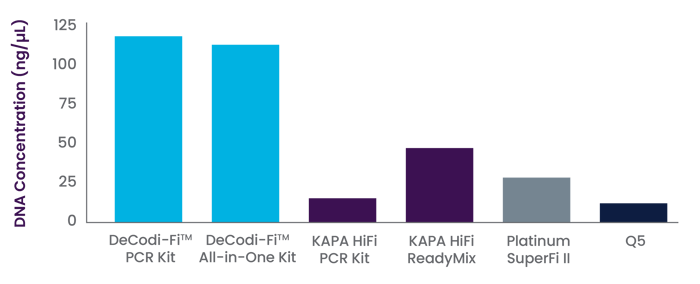
Concentración de ADN obtenida a partir de una reacción de PCR de 25 μL con un fragmento de lambda de 15 kb, utilizando DeCodi-Fi™ PCR kit, DeCodi-Fi™ All-in-One Mix y enzimas competidoras (KAPA HiFi, Platinum SuperFi II y Q5). El experimento se realizó por triplicado.
COBERTURA DE SECUENCIACIÓN +
Garantiza una amplificación con bajo sesgo, incluso al utilizar diferentes buffers y en fragmentos ricos en AT o GC, logrando una cobertura más uniforme y una mayor profundidad de secuenciación.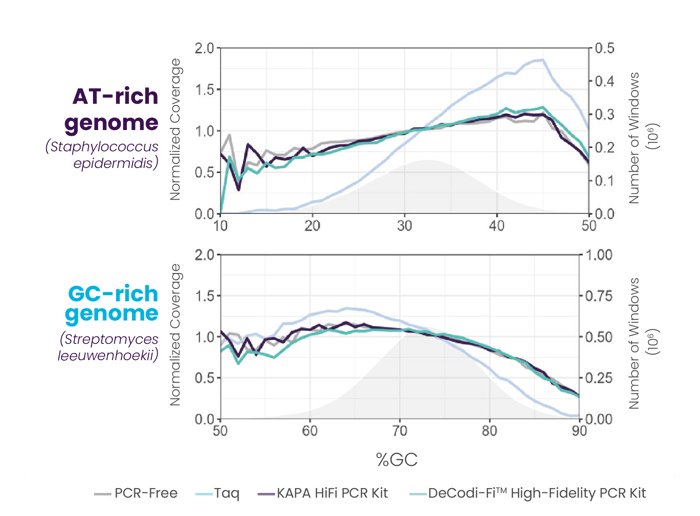
Las bibliotecas de Illumina preparadas a partir de un genoma común (Escherichia coli) fueron amplificadas utilizando Taq polymerase, KAPA HiFi PCR kit y DeCodi-Fi™ High-Fidelity PCR kit (izquierda). Las bibliotecas de Illumina preparadas a partir de un genoma rico en AT (Staphylococcus epidermidis) y un genoma rico en GC (Streptomyces leeuwenhoekii) fueron amplificadas utilizando Taq polymerase, KAPA HiFi PCR kit y DeCodi-Fi™ High-Fidelity PCR kit (derecha).
COMPATIBILIDAD CON PERLAS MAGNÉTICAS +
DeCodi-Fi 2X muestra mejor resistencia a perlas magnéticas en comparación con otros competidores, destacándose en PCR de amplicones largos. Esto convierte a DeCodi-Fi en una polimerasa de ADN de alta fidelidad confiable, robusta y de costo efectivo, ideal para diversas metodologías que requieren el uso de perlas magnéticas, sin comprometer el rendimiento.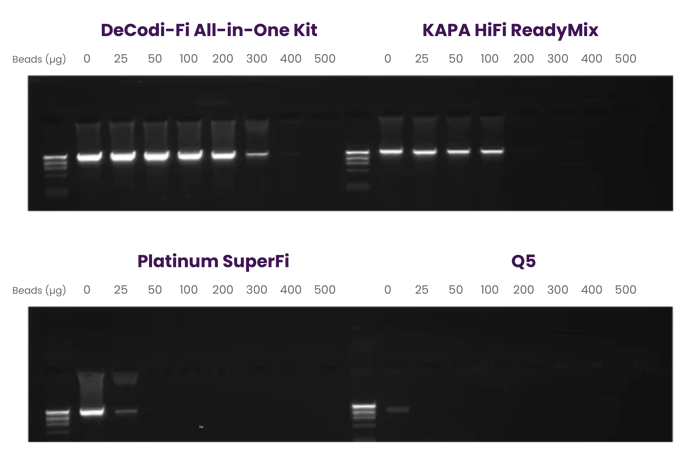
TOLERANCIA A URACILO +
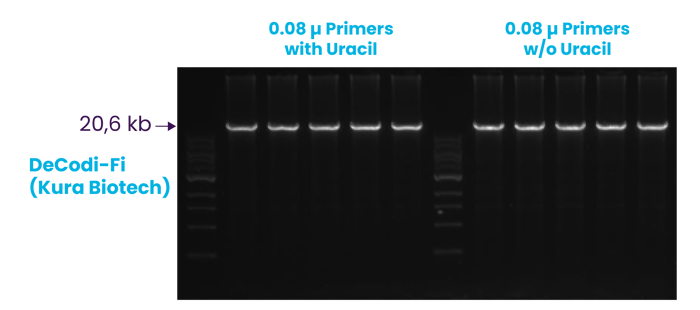
Amplificación robusta de un fragmento de ADN λ de 20,6 kb a partir de 2 ng de ADN inicial, realizada en pentaplicado, utilizando primers con y sin uracilo, lo que confirma una eficiencia de amplificación comparable y demuestra la tolerancia a primers que contienen uracilo.
ALTA FIDELIDAD +
DeCodi-Fi™ High-Fidelity Polymerase muestra una reducción del 64% en la tasa de errores en comparación con KAPA HiFi.

Comparación de DeCodi-Fi™, KAPA HiFi y Toyobo HiFi, medida en términos de mejora relativa sobre Taq Polymerase, basada en bibliotecas de Illumina del genoma completo de E. coli.
ALTO RENDIMIENTO +

Concentración de ADN obtenida a partir de una reacción de PCR de 25 μL con un fragmento de lambda de 15 kb, utilizando DeCodi-Fi™ PCR kit, DeCodi-Fi™ All-in-One Mix y enzimas competidoras (KAPA HiFi, Platinum SuperFi II y Q5). El experimento se realizó por triplicado.
COBERTURA DE SECUENCIACIÓN +
Garantiza una amplificación con bajo sesgo, incluso al utilizar diferentes buffers y en fragmentos ricos en AT o GC, logrando una cobertura más uniforme y una mayor profundidad de secuenciación.

Las bibliotecas de Illumina preparadas a partir de un genoma común (Escherichia coli) fueron amplificadas utilizando Taq polymerase, KAPA HiFi PCR kit y DeCodi-Fi™ High-Fidelity PCR kit (izquierda). Las bibliotecas de Illumina preparadas a partir de un genoma rico en AT (Staphylococcus epidermidis) y un genoma rico en GC (Streptomyces leeuwenhoekii) fueron amplificadas utilizando Taq polymerase, KAPA HiFi PCR kit y DeCodi-Fi™ High-Fidelity PCR kit (derecha).
COMPATIBILIDAD CON PERLAS MAGNÉTICAS +
DeCodi-Fi 2X muestra mejor resistencia a perlas magnéticas en comparación con otros competidores, destacándose en PCR de amplicones largos. Esto convierte a DeCodi-Fi en una polimerasa de ADN de alta fidelidad confiable, robusta y de costo efectivo, ideal para diversas metodologías que requieren el uso de perlas magnéticas, sin comprometer el rendimiento.

DATOS DE RENDIMIENTO
AMPLIFICACIÓN LARGA Y ALTO RENDIMIENTO +
Maximiza el rendimiento del fragmento objetivo con una amplificación altamente eficiente, incluso en fragmentos de hasta 44 kb de longitud.

Se realizó la amplificación de fragmentos de ADN lambda de 20 kb y 44 kb con DeCodi-Fi™ High-Fidelity Polymerase, KAPA HiFi, Platinum SuperFi II y Q5. Cada fragmento se amplificó a partir de una baja cantidad de ADN (1 ng). Las reacciones se llevaron a cabo bajo condiciones optimizadas para cada enzima, utilizando 24 ciclos. El experimento se realizó por triplicado.
ALTA FIDELIDAD +
DeCodi-Fi™ High-Fidelity Polymerase muestra una reducción del 64% en la tasa de errores en comparación con KAPA HiFi.

Comparación de DeCodi-Fi™, KAPA HiFi y Toyobo HiFi, medida en términos de mejora relativa sobre Taq Polymerase, basada en bibliotecas de Illumina del genoma completo de E. coli.
ALTO RENDIMIENTO +

Concentración de ADN obtenida a partir de una reacción de PCR de 25 μL con un fragmento de lambda de 15 kb, utilizando DeCodi-Fi™ PCR kit, DeCodi-Fi™ All-in-One Mix y enzimas competidoras (KAPA HiFi, Platinum SuperFi II y Q5). El experimento se realizó por triplicado.
COBERTURA DE SECUENCIACIÓN +
Garantiza una amplificación con bajo sesgo, incluso al utilizar diferentes buffers y en fragmentos ricos en AT o GC, logrando una cobertura más uniforme y una mayor profundidad de secuenciación.

Las bibliotecas de Illumina preparadas a partir de un genoma común (Escherichia coli) fueron amplificadas utilizando Taq polymerase, KAPA HiFi PCR kit y DeCodi-Fi™ High-Fidelity PCR kit (izquierda). Las bibliotecas de Illumina preparadas a partir de un genoma rico en AT (Staphylococcus epidermidis) y un genoma rico en GC (Streptomyces leeuwenhoekii) fueron amplificadas utilizando Taq polymerase, KAPA HiFi PCR kit y DeCodi-Fi™ High-Fidelity PCR kit (derecha).
COMPATIBILIDAD CON PERLAS MAGNÉTICAS +
DeCodi-Fi™ 2X muestra mejor resistencia a perlas magnéticas en comparación con otros competidores, destacándose en PCR de amplicones largos. Esto convierte a DeCodi-Fi™ en una polimerasa de ADN de alta fidelidad confiable, robusta y de costo efectivo, ideal para diversas metodologías que requieren el uso de perlas magnéticas, sin comprometer el rendimiento.

DOS FORMATOS DISPONIBLES
 DeCodi-Fi™
DeCodi-Fi™
2X All-In-One Mix
![]()
El formato All-in-One Mix ofrece nuestra polimerasa de alta fidelidad con HotStart en una configuración MasterMix 2X fácil de usar. Este formato incluye todos los componentes necesarios para la reacción (dNTPs, MgCl₂ y estabilizadores), excepto primers y molde, en un buffer propio.
Tipo de producto: All-in-One/MasterMix
Polimerasa: DeCodi-Fi™ HotStart High-Fidelity Polymerase
Formato: 5 mL
Reacciones: 400
Almacenamiento: Conservar a -20°C
 DeCodi-Fi™
DeCodi-Fi™
High-Fidelity PCR Kit
![]()
El High-Fidelity PCR Kit ofrece nuestra polimerasa de alta fidelidad con HotStart, junto con todos los componentes necesarios en formato separado. Incluye dos buffers: 5X High-Fidelity Buffer, recomendado para amplificar la mayoría de las secuencias con contenido balanceado de GC/AT y 5X GC-rich Buffer, recomendado para amplificar secuencias ricas en GC.
Tipo de producto: PCR kit
Polimerasa: DeCodi-Fi™ HotStart High-Fidelity Polymerase
Reacciones: 400
Almacenamiento: Conservar a -20°C
¿Necesitas una versión Non Hot-Start?
DeCodi-Fi™ también está disponible en una versión non hot-start, hecha a pedido y personalizable según tus necesidades.
DOS FORMATOS DISPONIBLES
 DeCodi-Fi™
DeCodi-Fi™
2X All-In-One Mix
![]()
El formato All-in-One Mix ofrece nuestra polimerasa de alta fidelidad con hotstart en una configuración MasterMix 2X fácil de usar. Este formato incluye todos los componentes necesarios para la reacción (dNTPs, MgCl₂ y estabilizadores), excepto primers y molde, en un buffer propio.
Tipo de producto: All-in-One/MasterMix
Polimerasa: DeCodi-Fi™ HotStart High-Fidelity Polymerase
Formato: 5 mL
Reacciones: 400
Almacenamiento: Conservar a -20°C
 DeCodi-Fi™
DeCodi-Fi™
High-Fidelity PCR Kit
![]()
El High-Fidelity PCR Kit ofrece nuestra polimerasa de alta fidelidad con hotstart, junto con todos los componentes necesarios en formato separado. Incluye dos buffers: 5X High-Fidelity Buffer, recomendado para amplificar la mayoría de las secuencias con contenido balanceado de GC/AT y 5X GC-rich Buffer, recomendado para amplificar secuencias ricas en GC.
Tipo de producto: PCR kit
Polimerasa: DeCodi-Fi™ HotStart High-Fidelity Polymerase
Reacciones: 400
Almacenamiento: Conservar a -20°C
Preguntas Frecuentes
DeCodi-Fi™
¿Cuál es el tiempo de extensión de DeCodi-Fi™? +
DeCodi-Fi™ generalmente requiere 30 segundos para extender fragmentos de ADN menores a 1 kilobase (kb). Para fragmentos más largos, agregar 15 segundos por cada kilobase adicional.
Por ejemplo, para amplificar un fragmento de 23 kb, el cálculo sería 30 segundos + (15 segundos × 22 kb) = 360 segundos (6 minutos).
¿Cuál es la cantidad de ADN recomendada para la reacción? +
DeCodi-Fi™ amplifica ADN de alta complejidad a partir de 0.1 ng y ADN de complejidad media desde 5 pg.
Cantidades recomendadas de entrada:
ADN genómico: 5–50 ng
Secuencias menores a 50 kb: menos de 1 ng
Para obtener amplificación en 25 ciclos, cargar más de 10⁴ copias del molde de ADN, sin exceder 100 ng por reacción (ejemplo: 35 ng de ADN genómico humano equivale a 10⁴ copias).
¿Cuáles son las condiciones de almacenamiento recomendadas para DeCodi-Fi™? +
Al recibir el kit, almacenar a -20°C inmediatamente para mantener su rendimiento óptimo y evitar ciclos repetidos de congelación y descongelación.
DeCodi-Fi™ está diseñada para ser transportada a temperaturas entre 2°C y 8°C sin afectar su rendimiento. Pruebas han confirmado que los componentes siguen siendo estables por al menos 10 días durante el transporte.
¿Cómo funciona la formulación HotStart? +
La polimerasa se mantiene inactiva por un anticuerpo hasta el ciclo inicial de desnaturalización térmica. Esto previene la hibridación no específica durante la preparación y el inicio de la reacción, reduciendo la amplificación no deseada y mejorando la eficiencia global de la PCR.
¿Cuántos ciclos se recomiendan para la amplificación? +
DeCodi-Fi™ tiene una eficiencia de amplificación superior a la de otras polimerasas comunes, incluso para amplicones largos.
Recomendaciones generales:
- PCR estándar (>10⁴ copias de plantilla): Comenzar con 25 ciclos.
- Amplicones >10 kb: No exceder 30 ciclos.
- Amplicones cortos: Pueden tolerar más ciclos sin afectar la calidad.
- Bibliotecas de secuenciación Illumina (<600 bp): 12-14 ciclos al partir de 1 ng de plantilla para alcanzar el rendimiento óptimo.
- Rango recomendado: Entre 10 y 30 ciclos, dependiendo de la secuencia, primers y aplicación.
Menos ciclos reducen la probabilidad de errores, minimizan productos no específicos, smearing y la acumulación de artefactos de alto peso molecular en geles de agarosa.
¿Ofrecen soluciones personalizadas? +
Sí, ofrecemos soluciones OEM y personalizadas que permiten aprovechar nuestra amplia experiencia en optimización de enzimas, refinamiento de buffers y fabricación.
¿Qué recomendaciones son importantes para el diseño de primers? +
Se recomienda agregar dos enlaces de fosforotioato en el extremo 3’ de los primers para evitar la degradación por exonucleasa 3’, debido a la fuerte actividad de proofreading de DeCodi-Fi™.
Esto es especialmente importante en:
- Primers cortos
- Amplificación de bibliotecas NGS
La degradación del extremo 3' del primer podría resultar en la amplificación de productos no específicos.
Símbolo en la síntesis de oligonucleótidos:
Las empresas de síntesis de oligos reconocen el asterisco (*) entre bases seleccionadas como indicación para ordenar un enlace de fosforotioato.
ejemplo de primer P5 con 2 enlaces de fosforotioato en el extremo 3’:
5'- AATGATACGGCGACCACCGA -3'
Preguntas Frecuentes
¿Cuál es el tiempo de extensión de DeCodi-Fi™? +
DeCodi-Fi™ generalmente requiere 30 segundos para extender fragmentos de ADN menores a 1 kilobase (kb). Para fragmentos más largos, agregar 15 segundos por cada kilobase adicional.
Por ejemplo, para amplificar un fragmento de 23 kb, el cálculo sería 30 segundos + (15 segundos × 22 kb) = 360 segundos (6 minutos).
¿Cuál es la cantidad de ADN recomendada para la reacción? +
DeCodi-Fi™ amplifica ADN de alta complejidad a partir de 0.1 ng y ADN de complejidad media desde 5 pg.
Cantidades recomendadas de entrada:
- ADN genómico: 5–50 ng
- Secuencias menores a 50 kb: menos de 1 ng
¿Cuáles son las condiciones de almacenamiento recomendadas para DeCodi-Fi™? +
Al recibir el kit, almacenar a -20°C inmediatamente para mantener su rendimiento óptimo y evitar ciclos repetidos de congelación y descongelación.
DeCodi-Fi™ está diseñada para ser transportada a temperaturas entre 2°C y 8°C sin afectar su rendimiento. Pruebas han confirmado que los componentes siguen siendo estables por al menos 10 días durante el transporte.
¿Cómo funciona la formulación HotStart? +
La polimerasa se mantiene inactiva por un anticuerpo hasta el ciclo inicial de desnaturalización térmica. Esto previene la hibridación no específica durante la preparación y el inicio de la reacción, reduciendo la amplificación no deseada y mejorando la eficiencia global de la PCR.
¿Cuántos ciclos se recomiendan para la amplificación? +
La polimerasa DeCodi-Fi™ tiene una eficiencia de amplificación superior a la de otras polimerasas comunes, incluso para amplicones largos.
Recomendaciones generales:
- PCR estándar (>10⁴ copias de plantilla): Comenzar con 25 ciclos.
- Amplicones >10 kb: No exceder 30 ciclos.
- Amplicones cortos: Pueden tolerar más ciclos sin afectar la calidad.
- Bibliotecas de secuenciación Illumina (<600 bp): 12-14 ciclos al partir de 1 ng de plantilla para alcanzar el rendimiento óptimo.
- Rango recomendado: Entre 10 y 30 ciclos, dependiendo de la secuencia, primers y aplicación.
Menos ciclos reducen la probabilidad de errores, minimizan productos no específicos, smearing y la acumulación de artefactos de alto peso molecular en geles de agarosa.
¿Ofrecen soluciones personalizadas? +
Sí, ofrecemos soluciones OEM y personalizadas que permiten aprovechar nuestra amplia experiencia en optimización de enzimas, refinamiento de buffers y fabricación.
¿Qué recomendaciones son importantes para el diseño de primers? +
Se recomienda agregar dos enlaces de fosforotioato en el extremo 3’ de los primers para evitar la degradación por exonucleasa 3’, debido a la fuerte actividad de proofreading de DeCodi-Fi.
Esto es especialmente importante en:
- Primers cortos
- Amplificación de bibliotecas NGS
La degradación del extremo 3' del primer podría resultar en la amplificación de productos no específicos.
Símbolo en la síntesis de oligonucleótidos:
Las empresas de síntesis de oligos reconocen el asterisco (*) entre bases seleccionadas como indicación para ordenar un enlace de fosforotioato.
ejemplo de primer P5 con 2 enlaces de fosforotioato en el extremo 3’:
5'- AATGATACGGCGACCACCGA -3'
¿Listo para amplificar tu investigación?
Lleva tu secuenciación de ADN al siguiente nivel con DeCodi-Fi™. Contacta a nuestro equipo para soporte técnico y ventas.
¿Listo para amplificar tu investigación?
Lleva tu secuenciación de ADN al siguiente nivel con DeCodi-Fi™.
Contacta a nuestro equipo para soporte técnico y ventas.